Denne underside hører til Biotech Academy’s gymnasie projekt Moderne Genteknologi
Teksten og billederne blev udarbejdet i forbindelse med Biotech Academy’s Camp 2020, og er blevet tilpasset projektet efterfølgende.


Teksten og billederne blev udarbejdet i forbindelse med Biotech Academy’s Camp 2020, og er blevet tilpasset projektet efterfølgende.
Uracil-Specific Excision Reagent (USER)-kloning er en metode til sammensætning af to eller flere DNA-sekvenser. Metoden bygger på brugen af Polymerase chain reaction (PCR) til at danne kompatible ender i sine DNA-sekvenser. Disse “sticky ends” kan skræddersyes, således at to eller flere DNA-sekvenser kan stykkes sammen med nærmest ethvert andet stykke DNA med lignende sticky ends. Typisk bruges USER-kloning til at indsætte en eller flere gen-sekvenser i et plasmid, for derefter at udtrykke genet i en passende værtscelle. Før at disse gener kan indsættes i plasmidet, skal plasmidet lineariseres med et eller flere restriktionsenzymer.
En typisk workflow med USER-kloning kan derfor opdeles i en række trin:
Med en PCR kan man tilføje overhangs til sit gene of interest (GOI, eller insert). For at danne disse overhangs, skal man udføre en PCR med primere, der indeholder “haler”, der ikke binder til ens GOI. Efter en gennemført PCR, vil ens PCR produkter indeholde overhangs, svarende til primernes haler.
I USER kloning indsætter man en bestemt sekvens i primer-halerne, der matcher med de sticky ends man senere laver i ens plasmid. Primer-sekvensen skal desuden indeholde nukleotiden uracil (U), som det niende nukleotid fra 5’ enden, som man kan se i figur 1. Uracil findes ikke naturligt i DNA, det findes til gengæld naturligt i RNA, hvor den indsættes på de pladser der normalt optages af thymin (T) i DNA. Thymins basepar er adenin; uracil kan derfor også danne basepar med adenin. Dette betyder at når man kører en PCR med uracil i primer-halen, vil den resulterende DNA-streng indeholde baseparet uracil-adenin figur.
For at fuldende USER kloning skal man fjerne uracilen for at danne de ønskede sticky ends, der matcher dem man har lavet i det lineariserede plasmid. Heldigvis er der enzymer der kan læse DNA, såsom USER enzymer. Hvis disse enzymer støder på et sted, hvor der er uracil på thymins plads, fjerner de uracilen. Når enzymet har fjernet uracilen fra DNA, vil de resterende 7 nukleotider fra DNA-fragmentet blive tabt. Når de 7 nukleotider går tabt, står man tilbage med et DNA-fragment med en sticky end i hver ende. Det er vigtigt at man designer sine primere til at indeholde den rigtige sekvens, der resulterer i sticky ends der er komplementære med det lineariserede plasmid.
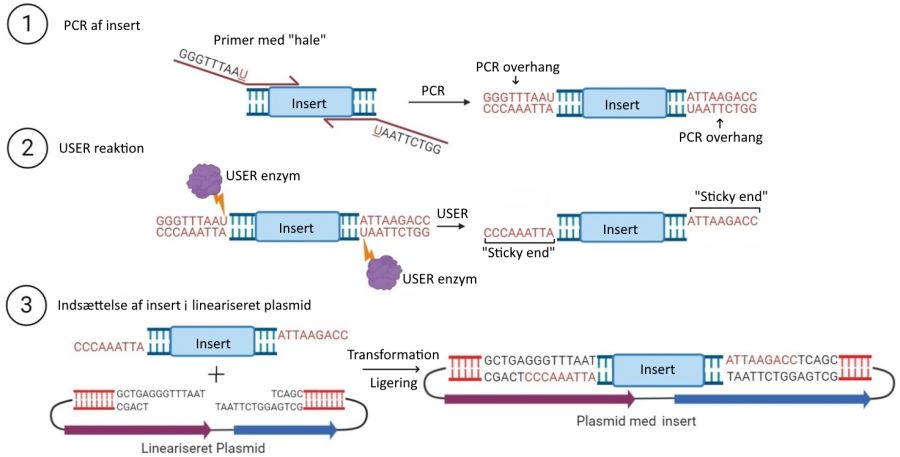
Figur 1. USER-kloning workflow. Ens ønskede gen amplificeres først med PCR, hvortil et sæt af primere med særlige haler er blevet designet. Produktet af PCR’en er ens gen, med et sæt af overhangs. Disse overhangs kan kløves af USER enzymet, hvilket producerer sticky ends. Er primerne designet korrekt, kan de producerede sticky ends bruges til at klone ens gen ind i et passende plasmid, der er blevet lineariseret med restriktionsenzymer. Efterfølgende kan det færdige plasmid transformeres ind i E. coli, som ligerer plasmidet, og laver kopier af plasmidet.
Plasmidet der skal bruges til en USER kloning skal indeholde et såkaldt USER cloning site (UCS). Et UCS består af tre genkendelses sites for to restriktionsenzymer. To af disse restriktionsenzymer er “nicking”-enzymer, hvorimod det tredie er et almindeligt restriktionsenzym, der kløver i begge DNA-strenge. Nicking enzymer derimod, kløver kun bindinger på en enkelt streng. En række af kombinationer af enzymer findes, og hvert laboratorie vil som regel have sin egen foretrukne metode. Som eksempel kan de to restriktionsenzymer PacI og Nt.BbvCI bruges. De tre genkendelses sites ligger ved siden af hinanden som afbilledet i Figur 2. PacI kløver DNAen over i to og Nt.BbvCI laver nicks på DNAen. Nukleotiderne der er placeret mellem den nye ende skabt af PacI og der hvor Nt.BbvCI har lavet et nick bliver tabt og man står tilbage med et linearliseret plasmid der indeholder kompatible sticky ends, som vist i Figur 1.
Figur 2. Linearisering af et plasmid, til brug i USER kloning. Det viste plasmid kløves af de to enzymer PacI og Nt.BbvCI, hvilket producerer sticky ends, der er kompatible med dem, produceret ved den tidligere nævnte PCR reaktion.
Efter man har brugt USER enzymet på sit insert og man har linearliseret sit plasmid, er man klar til at blande de to komponenter. Resultatet af at blande lineariseret plasmid med ens insert, er at man indsætter sit insert i ens plasmid, og på samme tid sørger for, at plasmidet får en cirkulær konformation igen. Det er dog ikke tilstrækkeligt, at plasmidet bare “lukker sig” om ens insert. For at fuldende kloningen skal plasmidet og ens insert fysisk kobles sammen i en proces kaldt ligering. I de ældre kloningsmetoder brugte man enzymet DNA ligase til dette formål, før man transformerede sit færdige plasmid, ind i en værtscelle. Nu til dags springer man ligeringstrinnet over, da bakterier kan udføre ligeringen for os. Man transformerer derfor blot sit ikke-ligerede plasmid ind i en bakterie, hyppigst E. coli, og lader bakterien ligere plasmidet. Herefter vil plasmidet befinde sig i værtscellen, og vil begynde at danne kopier af sig selv. Hvis plasmidet indeholder nye gener, vil værtscellen begynde at udtrykke disse.
Hvad du synes om Biotech Academy?