Cas9 og DNA identifikation
Denne underside udgør første del af teorien for Biotech Academys materiale om CRISPR-Cas9.
Genmodificering og DNA-dobbeltstrengsbrud


Denne underside udgør første del af teorien for Biotech Academys materiale om CRISPR-Cas9.
Cas9-proteinet danner præcise dobbeltstrengsbrud i selvvalgte positioner i DNA. Placeringen af dobbeltstrengsbruddet afhænger af det valgte gRNA, som er bundet i Cas9. Dobbeltstrengsbrud er oplagte muligheder for modificering af DNA-sekvensen. Det er enten muligt at indsætte sekvenser i dobbeltstrengsbrud eller lave mutationer i sekvensen, hvilket udføres ved udnyttelse af cellens egne DNA-reparationsmekanismer. Selve genmodifikationen ligger altså i, hvordan man udnytter dobbeltstrengsbrudene. Dobbeltstrengsbrudenes nøjagtige placering er helt essentiel for resultatet, da man gerne vil ramme specifikke gener eller regulatoriske sekvenser i DNA’et.
Dobbeltstrengsbrud i DNA er en meget alvorlig situation for en celle, hvis de ikke er skabt under cellens egne kontrollerede processer. Et dobbeltstrengsbrud adskiller genomet rent fysisk, idet begge DNA-strenge brydes, og kan have alvorlige konsekvenser, såsom celledød. Derfor vil cellen gøre alt for at samle DNA’et igen med sine reparationsmekanismer. De to hovedtyper af reparationssystemer er non-homologous end joining (NHEJ) og homology directed repair (HDR), der reparerer dobbeltstrengsbrud ved hver deres mekanisme.
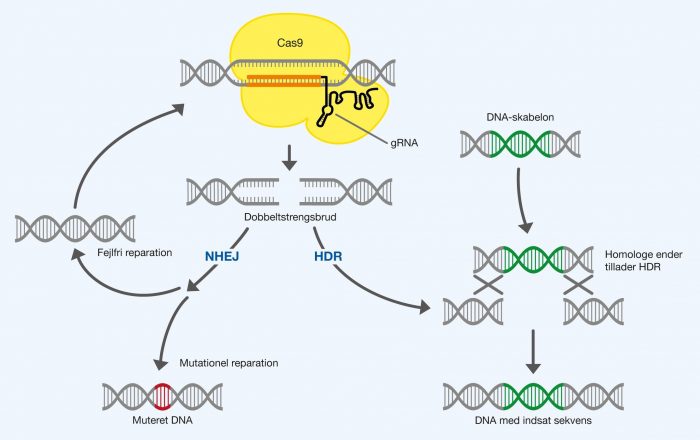
Figur 1. Cas9 danner dobbeltstrengsbrud i DNA ud fra placeringen valgt med det bundne gRNA. Dobbeltstrengsbrud repareres enten af non-homologous end joining (NHEJ) eller homology directed repair (HDR). Disse reparerer DNA ved to forskellige mekanismer og derfor kan man opnå forskellige resultater af reparationen. NHEJ samler enderne af DNA, men kan begå fejl, der danner mutationer. Foregår NHEJ korrekt, vil Cas9 blot kunne genkende DNA-sekvensen igen og danne et nyt brud. Dette gentager sig indtil en mutation er dannet. HDR er anderledes, da mekanismen anvender en DNA-skabelon til at reparere bruddet. DNA-skabelonen er normalt en kopi af den originale sekvens. Dog kan man selv bestemme indholdet af DNA-skabelonen, ved at snyde reparationssystemet, hvormed man kan indsætte selvvalgte DNA-sekvenser. Dette kræver at DNA-skabelonen har homologe ender til dobbeltstrengsbruddet. Man udnytter disse to reparationssystemer til genmodificering i den position, hvor man har placeret sit dobbeltstrengsbrud.
Non-homologous end joining (NHEJ) er en reparationsmekanisme, der sammensætter de to løse ender af DNA-strengene efter et dobbeltstrengsbrud. DNA-enderne sammensættes af enzymet DNA ligase IV i mennesker. Denne reparationsmekanisme er meget effektiv, men kan forårsage mutationer. Disse mutationer kan både være insertioner (indsættelse) og deletioner (fjernelse) af basepar, som begge har potentiale til at forårsage frame-shift mutationer. Proteinkodende DNA aflæses som codons, som er intervaller af 3 nukleotider. En ændring der ikke følger dette system vil forstyrre fortolkningen af koden. DNA-læserammen forskydes af frame-shift mutationer, da der indsættes eller fjernes basepar i et antal, der ikke er et multiplum af 3. Det kan også ske, at frame-shift mutationen tilfældigvis indfører et stop-codon i koden, der stopper proteintranslationen utidigt. Mutationerne betyder, at DNA-sekvensen ikke længere kan aflæses korrekt og at proteinet, som sekvensen koder for, vil blive dysfunktionelt.
Reparationen er sandsynligvis succesfuld i de fleste tilfælde. Den vigtige pointe er her, at Cas9 kan genkende den reparerede sekvens igen og lave et nyt dobbeltstrengsbrud, som igen kan blive repareret af NHEJ. Denne proces vil gentage sig, indtil NHEJ fejlagtigt laver en mutation i DNA-sekvensen. Så vil Cas9 ikke længere kunne genkende DNA-sekvensen, da den er permanent ændret med en mutation. Det smarte er, at mutationen er blevet indført på positionen, som var bestemt ved det selvvalgte gRNA. På denne måde kan man anvende Cas9 til dannelse af permanente mutationer i selvvalgte positioner i en DNA-sekvens. Dette er illustreret på figur 1.
Homology directed repair (HDR) er en mere nøjagtig og kompliceret reparationsmekanisme, der anvender en DNA-sekvens som skabelon til at reparere et dobbeltstrengsbrud. Her indsættes en DNA-sekvens i bruddet, som er lig den anvendte skabelon. DNA-skabelonen skal have ender, der er homologe til enderne af dobbeltstrengsbruddet. Det betyder, at enderne har overlappende og ens DNA-sekvenser mellem de to stykker DNA. Dette er smart til den naturlige brug af reparationssystemet, da der ofte findes flere kopier af de samme DNA-sekvenser i cellen. Herved kan et stykke identisk DNA findes ud fra de homologe ender og fungere som skabelon til genskabelsen af den originale sekvens efter et brud.
HDR kan kun basere sin reparation på baggrund af de homologe ender af DNA-skabelonen, men ikke hvad der er imellem. Derfor kan man tvinge reparationssystemet til at indsætte selvvalgte DNA-sekvenser, så længe de har homologe ender. Når man vil indsætte en sekvens i en bestemt position i DNA’et, danner man først dobbeltstrengsbruddet med sit Cas9. Ved derefter at indføre en kunstig DNA-skabelon med homologe ender, kan man indsætte sekvensen i bruddet ved udnyttelse af reparationsmekanismen. Dette er illustreret på figur 1.
Kort opsummeret kan det siges, at NHEJ kan samle DNA-enderne og eventuelt indføre små tilfældige mutationer. HDR kan genoprette den originale DNA-sekvens eller indføre en ny DNA-sekvens afhængigt af en DNA-skabelon med homologe ender til dobbeltstrengsbrudet.
CRISPR/Cas9-genmodificeringssystemet behøver kun to komponenter for at være aktivt, nemlig Cas9-proteinet og det valgte gRNA.
Cas9-proteinet målsøger specifikke DNA-sekvenser ved at bruge gRNA, som er bundet i Cas9 og kan udskiftes. Dette gRNA har en sekvens på 20 nukleotider i den ene ende, der er komplementær til den målsøgte DNA-sekvens, som tilsvarende også er 20 nukleotider lang.
De 20 nukleotider i gRNA genkender DNA-sekvenserne, ved at binde sig til dem og derfor bestemmer gRNA, hvilken DNA-sekvens Cas9 eftersøger. På denne måde kan man omprogrammere Cas9 til at genkende forskellige DNA-sekvenser, ved at vælge sekvensen af det bundne gRNA.
Der findes et yderligere krav udover gRNA-sekvensen. Den målsøgte DNA-sekvens skal indeholde en PAM-sekvens. Denne er 5’-NGG-3’ for Cas9 fra S. pyogenes, hvor den første base (N) kan være enhver base, efterfulgt af 2 guanin-baser i 5’ til 3’ retningen af DNA-strengen.
PAM-sekvensen ligger altid på den modsatte streng end den streng, hvorpå de 20 nukleotider, der genkendes, ligger. De 20 nukleotider skal ligge i direkte forlængelse af PAM-sekvensen.
På figur 2 ses hvordan kan målsøge forskellige DNA-sekvenser, ved udskiftning af gRNA i Cas9 og placeringen af PAM-sekvensen i forhold til de målsøgte 20 nukleotider.
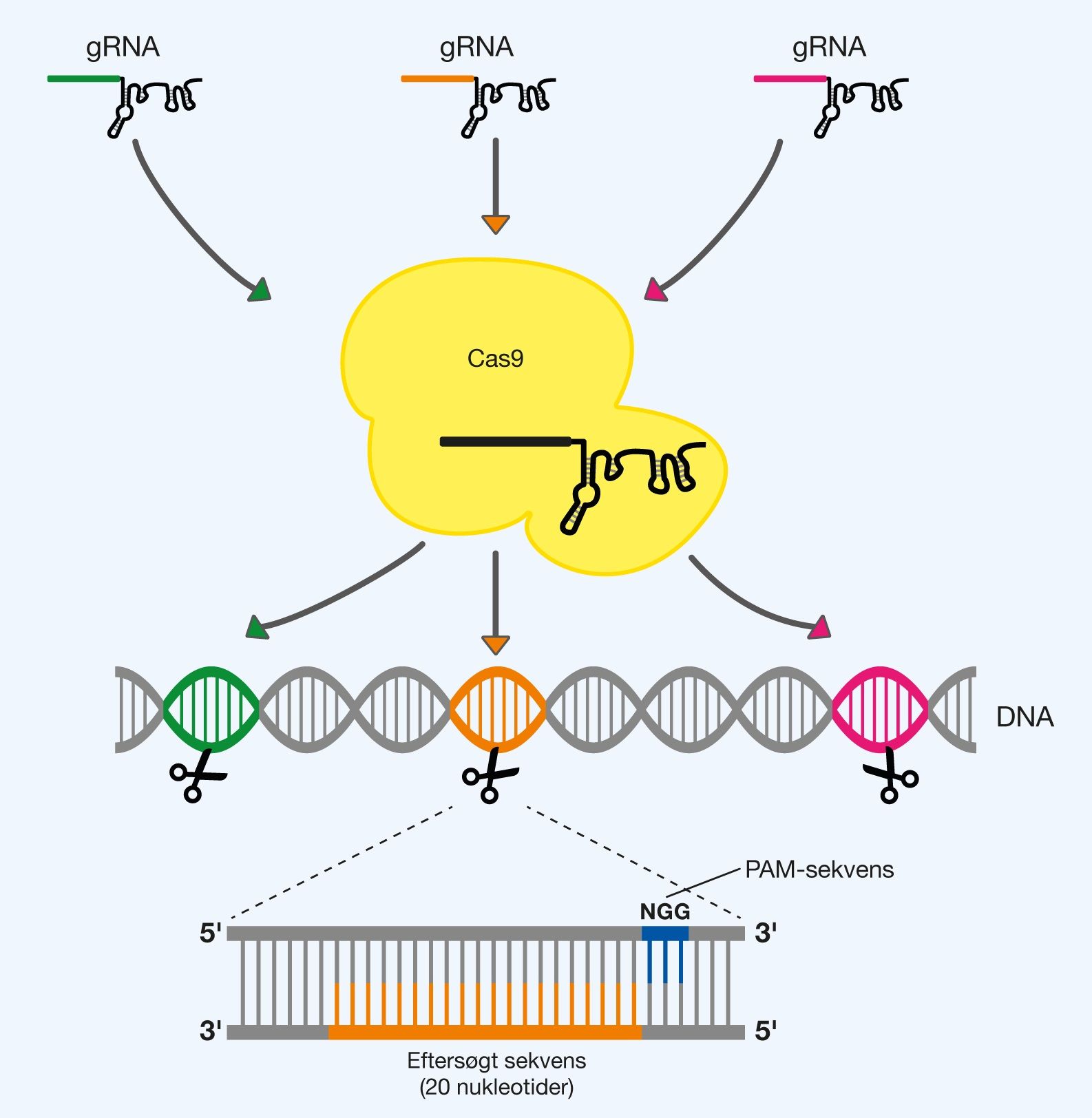
Figur 2. Omprogrammering af Cas9 med forskellige gRNA. Cas9-proteinet genkender forskellige DNA-sekvenser alt efter de 20 nukleotider i det bundne gRNA. Der skal være en 5′-NGG-3′ PAM-sekvens tilstede på den modsatte streng ved siden af de 20 eftersøgte nukleotider, for at genkendelsen af DNA-sekvensen kan finde sted.
Proteinet består af to hoveddele, identifikationsdelen (REC), der er ansvarlig for identifikationen af den specifikke DNA-sekvens og nukleasedelen (NUC), som står for kløvningen af DNA-sekvensen. Et PAM-interagerende (PI) domæne sidder i NUC og er delen af Cas9, som genkender PAM-sekvensen 5’-NGG-3’. REC fastholder gRNA i den ene ende, således at de genkendende 20 nukleotider i den anden ende er blottet på overfladen af Cas9, hvilket muliggør identifikationen af den specifikke DNA-sekvens. Når gRNA har bundet sig til det eftersøgte DNA, befinder det sig i grænsefladen mellem REC og NUC, hvilket kan ses på figur 3.
At Cas9 er en endonuklease, betyder at den kan spalte inde midt i DNA og ikke blot i enderne. I NUC haves to endonuklease-domæner, der kløver hver deres streng af det eftersøgte DNA. HNH-domænet kløver den DNA-streng, som indeholder sekvensen, der eftersøges af gRNA. RuvC-domænet kløver den modsatte DNA-streng, som indeholder PAM-sekvensen. Til sammen danner de to endonukleaser det præcise dobbeltstrengsbrud i DNA’et.
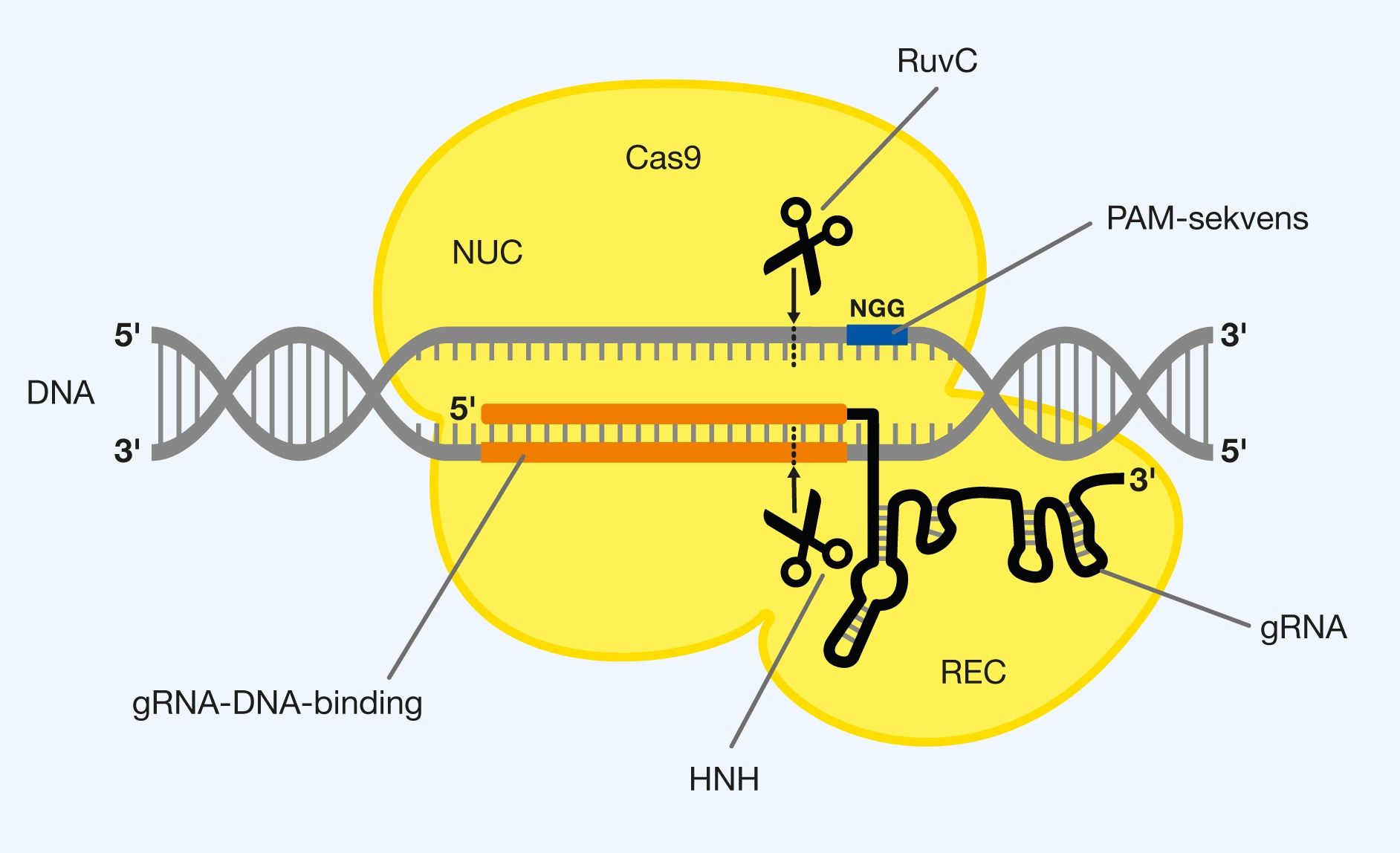
Figur 3. Strukturen af Cas9 med gRNA. Cas9 består af 2 hoveddele, kaldet REC og NUC. gRNA er bundet i REC, der står for identifikation af DNA-sekvenser. NUC indeholder et domæne, der genkender PAM-sekvensen (5’-NGG-3’). NUC står for kløvningen af DNA og har 2 endonuklease-domæner, hvoraf RuvC klipper DNA-strengen med PAM-sekvensen og HNH klipper DNA-strengen, der genkendes af gRNA. Dobbeltstrengsbruddet dannes mellem 3. og 4. bp efter PAM-sekvensen, indeni den genkendte sekvens.
Denne komplekse proteinstruktur udgør altså et biokemisk apparat, der er designet til at identificere DNA-sekvenser nøjagtigt, og derefter spalte dem præcist.
Nu da Cas9 proteinstrukturen er beskrevet, kan der tages et nærmere blik på hvordan de enkelte dele udnyttes i dannelsen af et dobbeltstrengsbrud i en specifik DNA-sekvens. Denne interaktion er meget vigtigt for Cas9, da det er den som man udnytter, når man bruger Cas9 som et genteknologisk værktøj. De følgende trin beskriver identifikationen og spaltningen af DNA, samt de krav der skal opfyldes, for at det er muligt.
Opsummering af Cas9-interaktion med DNA
De vigtigste informationer for dannelsen af dobbeltstrengsbrud med Cas9 kan ses på figur 4.
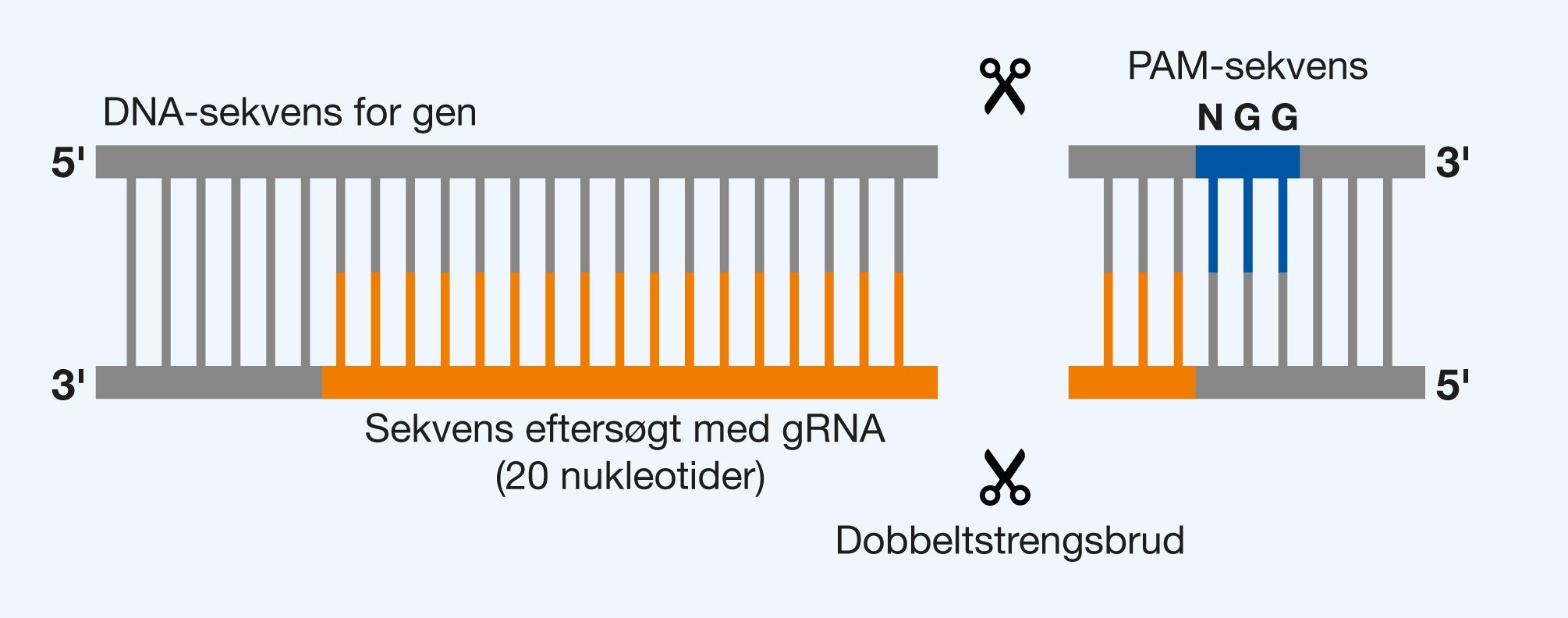
Figur 4. De vigtige elementer for Cas9 i genkendelsen og kløvningen af en specifik DNA-sekvens. Tilstedeværelsen af PAM-sekvensen 5’-NGG-3’ tillader, at de efterfølgende 20 nukleotider på den modsatte streng kan genkendes af det bundne gRNA i Cas9. Cas9 danner dobbeltstrengsbruddet mellem det 3. og 4. basepar efter PAM-sekvensen indeni den genkendte sekvens.
Man kan anvende Cas9 på mange forskellige måder, som resulterer i forskellige molekylære ændringer af DNA-sekvensen. Der vil nu tages et nærmere blik på nogle af de grundlæggende strategier, som værktøjet kan bruges til og hvordan de resulterende DNA-sekvenser vil se ud. De forskellige strategier afhænger af, hvordan man har designet sit gRNA og hvilken reparationsmekanisme man udnytter. Ved at variere, hvilke komponenter man anvender, kan forskellige resultater af genmodifikationen opnås. Det er eksempelvis muligt at udnytte flere stykker gRNA. Denne taktik kaldes multiplexing og tillader, at man kan modificere flere DNA-sekvenser på én gang eller klippe store stykker DNA ud. Tilføjer man en DNA-skabelon, kan man opnå indsættelser af DNA-sekvenser.
De molekylære strategier defineres af, hvordan DNA-sekvensen bliver påvirket.
Destruktion
Ødelæggelse af genstrukturen ved små insertioner eller deletioner (samlet kaldet indels) i DNA-sekvensen. Cas9 anvendes med gRNA til at udvælge lokationen for dobbeltstrengsbruddet, hvorefter en indel opstår som resultat af mutationel NHEJ-reparation af bruddet. Multiplexing med flere stykker gRNA kan anvendes til at lave flere destruktioner af det samme gen eller forskellige gener på én gang. Se figur 5.
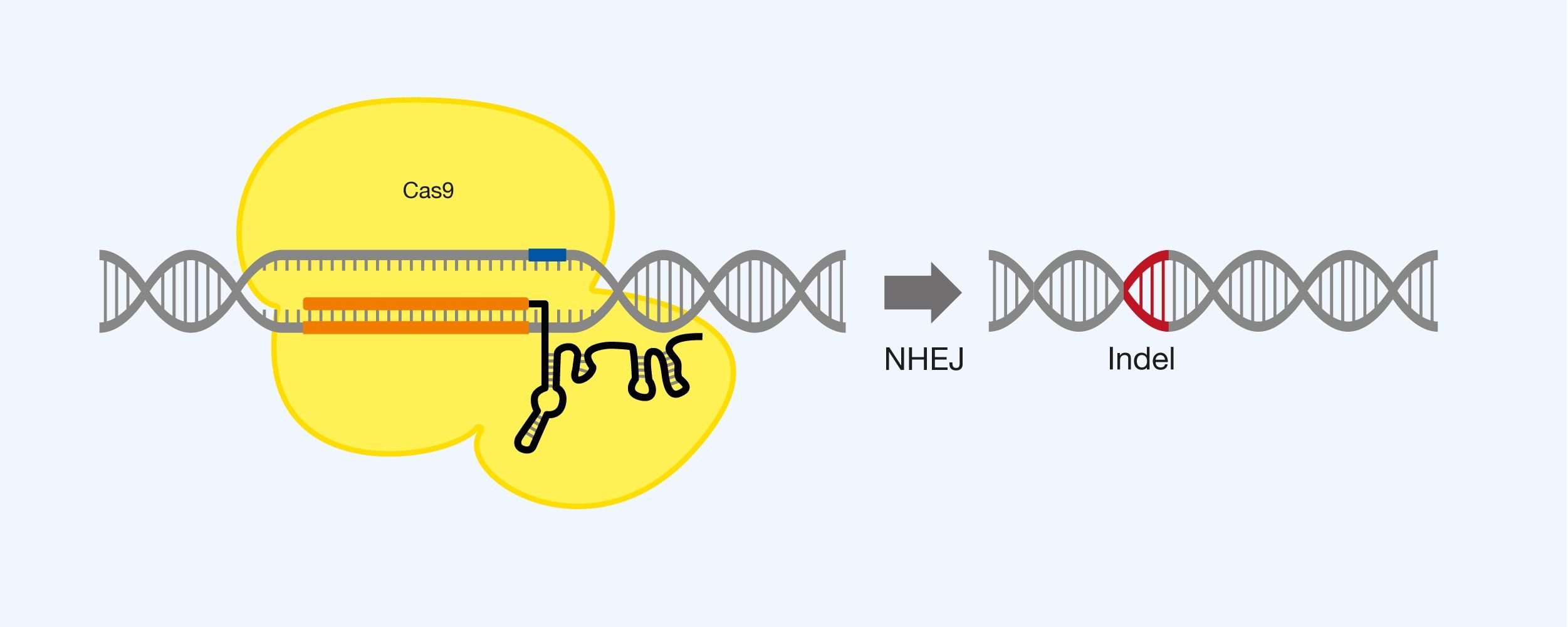
Figur 5. Destruktion af en DNA-sekvens ved brug af Cas9 og gRNA, samt udnyttelse af non-homologous end joining (NHEJ).
Insertion
Indsættelse af større sekvenser i genomet. Cas9 anvendes sammen med ét gRNA til at finde indsættelsespunktet, yderligere indføres en DNA-skabelon til indsættelse ved udnyttelse af HDR. Indsættes sekvensen midt i en anden sekvens, vil denne sekvens sandsynligvis ødelægges. Se figur 6.
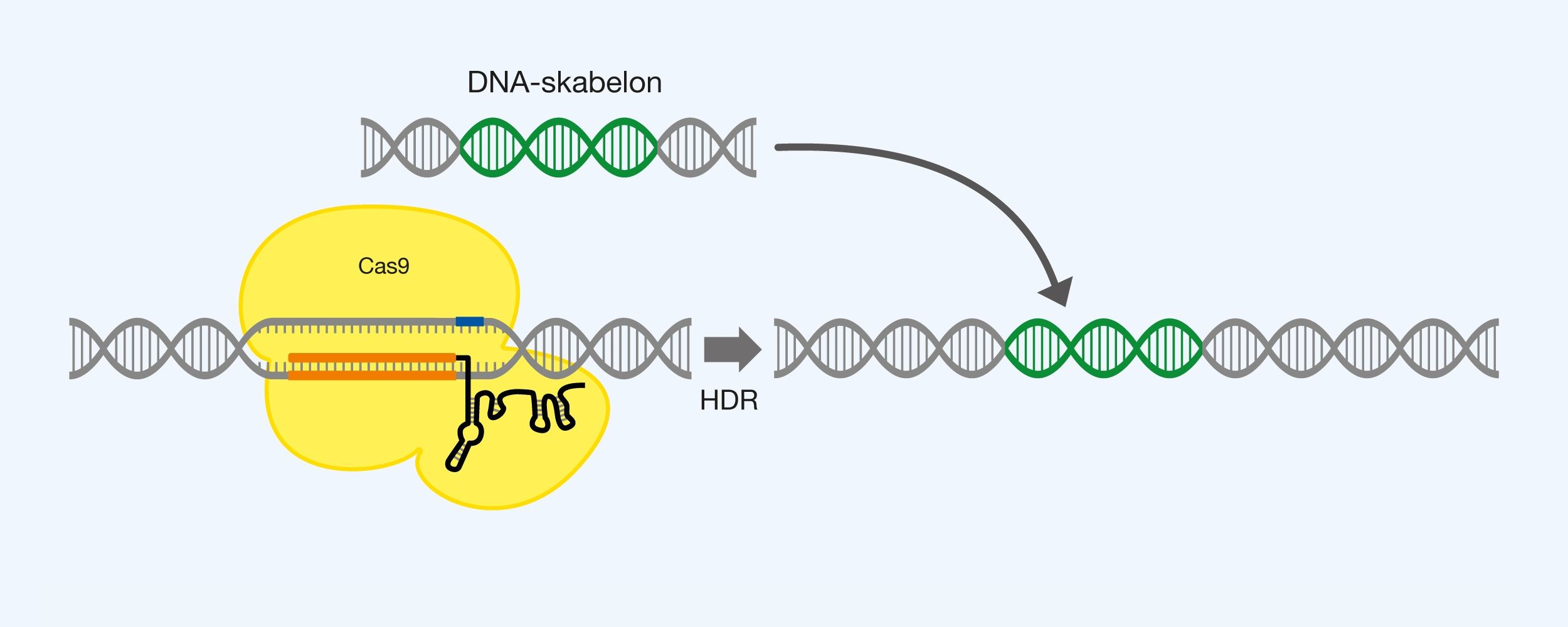
Figur 6. Insertion af en DNA-sekvens ved brug af Cas9, gRNA og en DNA-skabelon, samt udnyttelse af homology directed repair (HDR).
Excision
Deletion af større sekvenser. Cas9 anvendes sammen med to stykker gRNA, der hver især markerer endepunkterne for den sekvens man vil fjerne. Efter excisionen af sekvensen, kan det større brud eksempelvis repareres ved NHEJ, der samler de to endepunkter. Se figur 7.
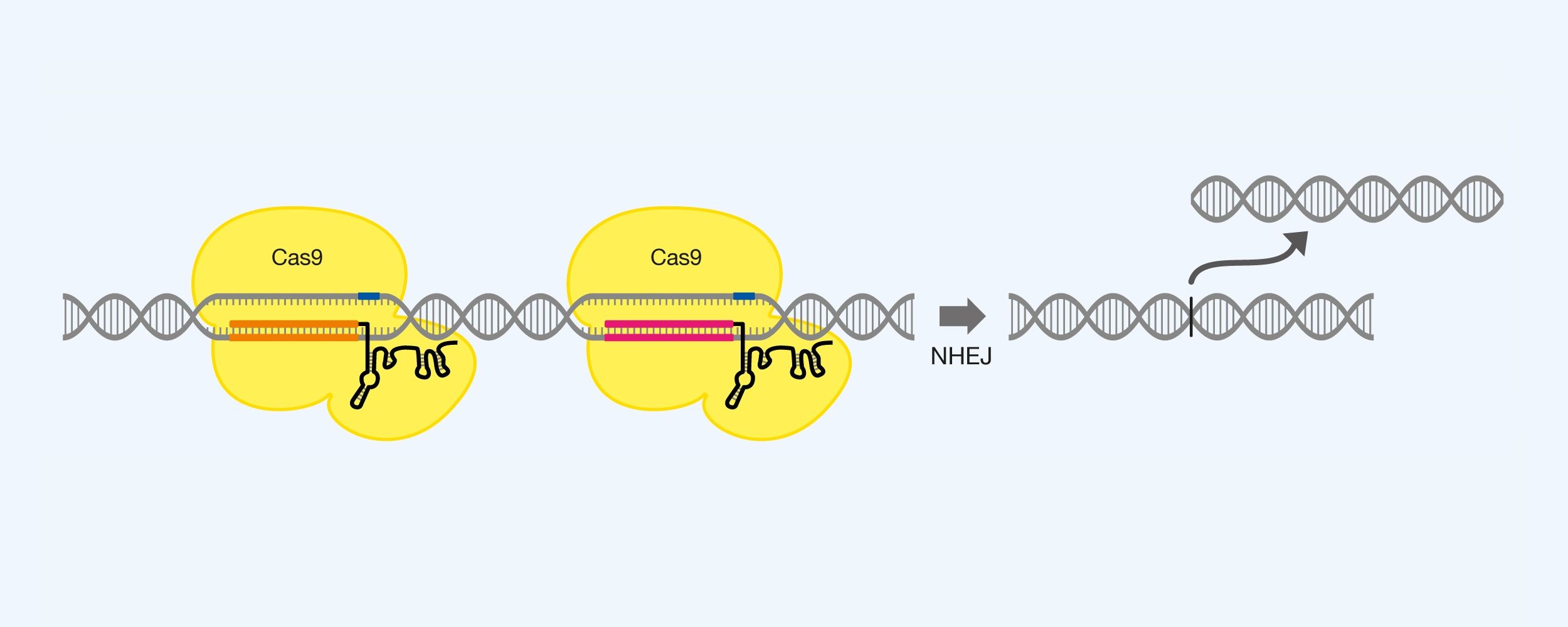
Figur 7. Excision af en DNA-sekvens ved brug af Cas9 og to stykker gRNA, samt udnyttelse af non-homologous end joining (NHEJ).
Erstatning
Større sekvens substitueres med en anden sekvens. Cas9 anvendes med to gRNA, der hver især markerer endepunkterne for sekvensen der skal erstattes, som gjort ved excision. En DNA-skabelon indføres, ligesom ved insertion, der indsættes i det større brud ved udnyttelse af HDR. Se figur 8.
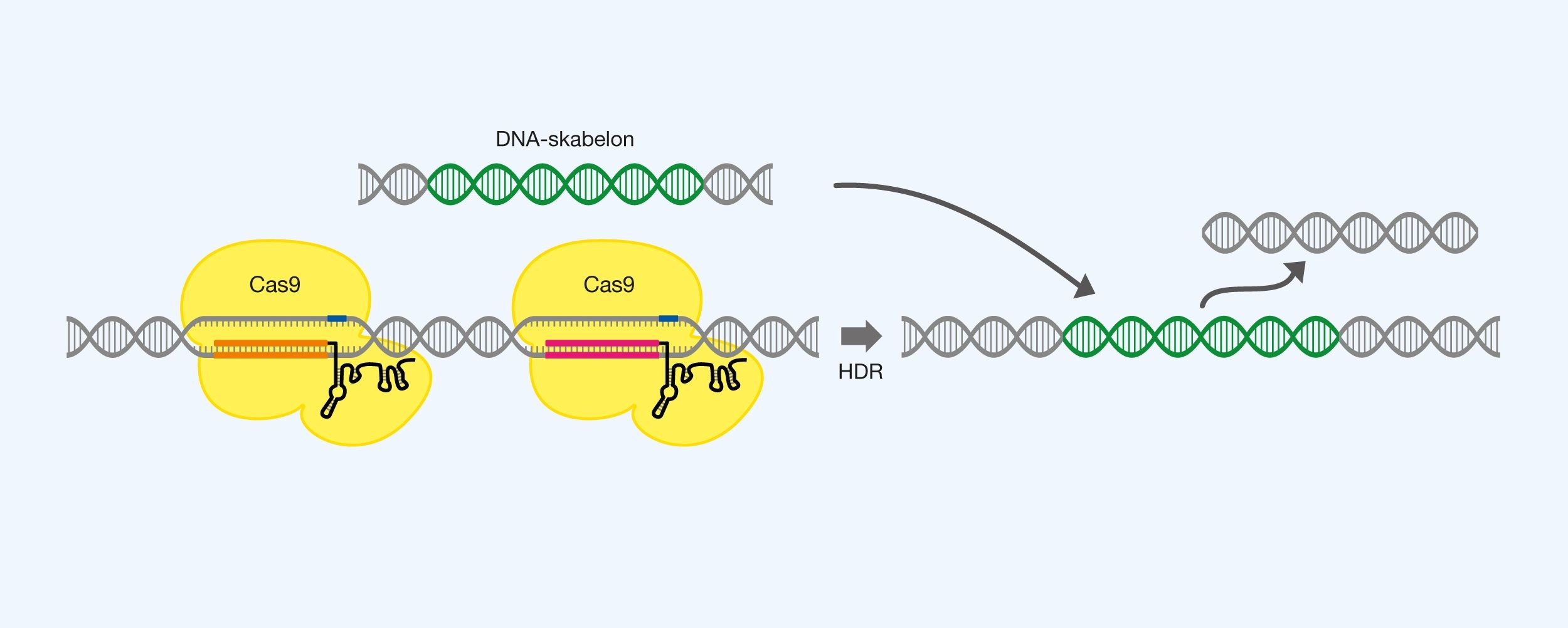
Figur 8. Erstatning af en DNA-sekvens ved brug af Cas9, to stykker gRNA og en DNA-skabelon, samt udnyttelse af homology directed repair (HDR).