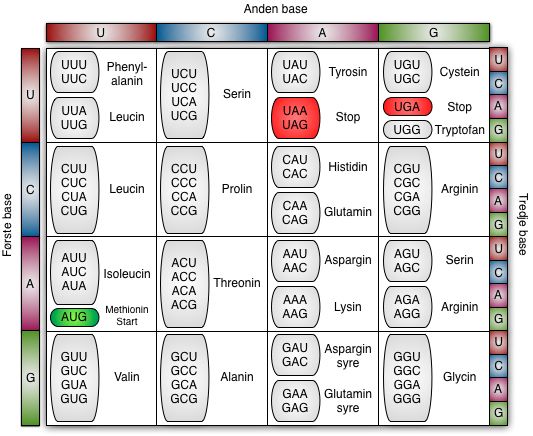
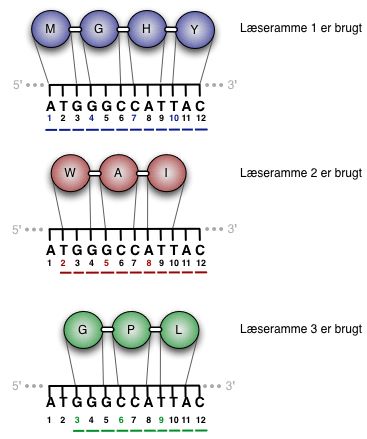
Læserammer
Da DNA-sekvenser aflæses i codons (tre nukleotider) og ikke én nukleotid ad gangen, kan der dannes forskellige aminosyrekæder alt afhængig af, i hvilken position på DNA-sekvensen translationen starter. Man siger, at DNA-sekvensen har forskellige læserammer. Antallet af læserammer (forskellige aminosyresekvenser), der kan dannes pr. DNA-streng, er dog begrænset til tre. Grunden til dette er, at en codon som nævnt består af tre nukleotider, og dermed aflæses DNA-sekvensen tre nukleotider ad gangen. Hvis man starter translationen på position 1 eller position 4, vil man således benytte den samme læseramme, og man vil dermed få den samme aminosyresekvens (dog uden den første aminosyre, hvis man starter på position 4). Den samme aminosyrekæde vil fås, hvis man starter på position 7, blot uden de to første aminosyrer etc. Hvis man starter på position 2, 5, 8… vil man få aminosyresekvensen fra læseramme 2. Denne aminosyresekvens vil være en anden end den fra læseramme 1, hvor man startede på position 1, 4, 7… Endelig er det samme princip gældende for læseramme 3, hvor aminosyresekvensen bliver translateret fra position 3, 6, 9…, se figur 2.
Da DNA-molekyler er sat sammen af to DNA-strenge i en dobbeltbundet DNA-helix, findes der ikke kun tre læserammer, men faktisk seks forskellige læserammer for hvert DNA-molekyle; tre forskellige læserammer på hver DNA-streng.
Læserammen mellem to stopcodons betegnes den åbne læseramme (eng. Open Reading Frame, ORF) og er den del af DNA’et, der kan kode for et protein.